EasyPGX®: la qPCR liofila che rivoluziona la lotta contro il cancro
EasyPGX® è la soluzione completa per la diagnostica in vitro, basata su RT-PCR, con il portfolio più ampio di test dedicato all’oncologia molecolare. Con il suo rivoluzionario formato reagenti anidro, pronto all’uso e pre aliquotato in strip di reazione da 8 pozzetti, EasyPGX® è adatto a tutte le routine di laboratorio, garantendo le migliori performance e risultati standardizzati in tempi rapidi.
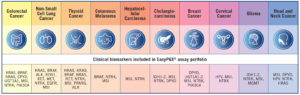
Caratteristiche principali:
- Pronto all’uso: reagenti in formato anidro, pre-aliquotati in strip da 8 pozzetti.
- Semplice da utilizzare: riduzione degli step di pipettamento con un hands-on-time < 10 minuti. Nessuna necessità di congelamento, scongelamento o pipettamento sul ghiaccio.
- Elevata sensibilità: limite di rilevamento fino allo 0,5%.
- Ampia flessibilità di campione: validato a partire da basse quantità di DNA, ctDNA e RNA da varie tipologie di matrici, tra cui FFPE e biopsia liquida.
- Veloce: dal tessuto al risultato in meno di 3 ore.
- Flessibile: possibilità di eseguire differenti target in un unico esperimento.
- Analisi automatica dei dati: il sistema include un software dedicato per l’analisi e l’interpretazione dei risultati.
- Trasporto e stoccaggio a temperatura ambiente: master mix complete in formato liofilo, stabili a temperatura ambiente.
- Standard inclusi: controlli positivi e negativi inclusi per la validazione di ogni sessione di analisi.
- Certificazione: i kit sono conformi alla direttiva EU CE, IVDR (2017/746).
EasyPGX system workflow: dal tessuto al risultato in meno di 3 ore.

Caratteristiche
Rilevamento delle principali mutazioni dell’esone 2 (codoni 12, 13), dell’esone 3 (codoni 59, 61), dell’esone 4 (codoni 117, 146) del gene KRAS mediante 8 miscele di oligonucleotidi. Ogni miscela di oligonucleotidi permette la co-amplificazione di uno o più alleli mutati e di un gene di controllo endogeno.
Una specifica miscela di oligonucleotidi di controllo consente la valutazione della qualità e della quantità del DNA presente nei campioni.
Controlli
- Controllo positivo, contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Sensibilità
Il kit permette di rilevare basse percentuali di allele mutato in presenza di elevate quantità di DNA genomico wild-type mediante amplificazione real-time con sonde sequenza specifiche marcate con FAM ed HEX. LOD fino a 0,5%.
Campione di partenza
ll kit permette l’analisi di DNA estratto da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e plasma*.
*Reagenti, consumabili e strumenti accessori per estrazione da plasma sono venduti separatamente (vedere linea Helix, codice H8040).
Caratteristiche
Rilevamento delle principali mutazioni del codone 600 di BRAF mediante 4 miscele di oligonucleotidi.
Ogni miscela di oligonucleotidi permette la co-amplificazione di uno o più alleli mutati e di un gene di controllo endogeno.
Una specifica miscela di oligonucleotidi di controllo consente la valutazione della qualità e della quantità del DNA presente nei campioni.
Controlli
- Controllo positivo, contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Sensibilità
Il kit permette di rilevare basse percentuali di allele mutato in presenza di elevate quantità di DNA genomico wild-type mediante amplificazione real-time con sonde sequenza specifiche marcate con FAM ed HEX. LOD fino a 0,5%.
Campione di partenza
ll kit permette l’analisi di DNA estratto da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e plasma*.
*Reagenti, consumabili e strumenti accessori per estrazione da plasma sono venduti separatamente (vedere linea Helix, codice H8040).
Caratteristiche
Rilevamento delle principali mutazioni, inserzioni, delezioni degli esoni 18, 19, 20, 21 di EGFR mediante 8 miscele di oligonucleotidi.
Ogni miscela di oligonucleotidi permette la co-amplificazione di uno o più alleli mutati e di un gene di controllo endogeno.
Una specifica miscela di oligonucleotidi di controllo consente la valutazione della qualità e della quantità del DNA presente nei campioni.
Controlli
- Controllo positivo, contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Sensibilità
Il kit permette di rilevare basse percentuali di allele mutato in presenza di elevate quantità di DNA genomico wild-type mediante amplificazione real-time con sonde sequenza specifiche marcate con FAM ed HEX. LOD fino a 0,5%.
Campione di partenza
Il kit permette l’analisi di DNA estratto da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e plasma*.
*Reagenti, consumabili e strumenti accessori per estrazione da plasma sono venduti separatamente (vedere linea Helix, codice H8040).
Caratteristiche
Rilevamento delle principali mutazioni dell’esone 2 (codoni 12, 13), dell’esone 3 (codoni 59, 61), dell’esone 4 (codoni 117, 146) del gene NRAS mediante 8 miscele di oligonucleotidi. Ogni miscela di oligonucleotidi permette la co-amplificazione di uno o più alleli mutati e di un gene di controllo endogeno. Una specifica miscela di oligonucleotidi di controllo consente la valutazione della qualità e della quantità del DNA presente nei campioni.
Controlli
- Controllo positivo, contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Sensibilità
Il kit permette di rilevare basse percentuali di allele mutato in presenza di elevate quantità di DNA genomico wild-type mediante amplificazione real-time con sonde sequenza specifiche marcate con FAM ed HEX. LOD fino a 0,5%.
Campione di partenza
ll kit permette l’analisi di DNA estratto da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e plasma*.
*Reagenti, consumabili e strumenti accessori per estrazione da plasma sono venduti separatamente (vedere linea Helix, codice H8040)
Caratteristiche
Rilevamento delle principali traslocazioni cromosomiche che coinvolgono i geni ALK, ROS1, RET e la perdita dell’esone 14 nel gene MET. Ogni miscela di oligonucleotidi permette la co-amplificazione di una o più fusioni e di un gene di controllo endogeno.
Controlli
- Controllo positivo, contiene una miscela di sequenze di RNA-DNA sintetico che corrispondono alle principali fusioni rilevate dal kit;
- Controllo negativo.
Materiale di partenza
RNA da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e campioni citologici.
Caratteristiche
Rilevamento, mediante discriminazione allelica, dei cinque polimorfismi DPYD*2A (IVS14+1G>A, c.1905+1G>A, rs3918290), DPYD*13 (c.1679T>G, rs55886062), DPYD D949V (c.2846A>T, rs67376798), DPYD IVS10 (c.1129– 5923C>G, rs75017182), DPYD*6 (c.2194G>A, rs1801160) del gene DPYD associati alla tossicita’ al trattamento con fluoropirimidine mediante 4 miscele di oligonucleotidi. Ogni miscela di oligonucleotidi permette la co-amplificazione delle sequenze mutate (FAM) e di quelle wild-type (HEX).
Controlli
- DPYD WT positive control: controllo positivo contenente una miscela di DNA sintetico wild-type per tutti i polimorfismi DPYD analizzati;
- DPYD MT positive control: controllo positivo contenente una miscela di DNA sintetico mutato per tutti i polimorfismi DPYD analizzati;
- Controllo negativo.
Campione di partenza
Il kit permette l’analisi di DNA genomico estratto da sangue intero.
Caratteristiche
Rilevamento, mediante discriminazione allelica, dei polimorfismi UGT1A1*1 (TA)6, UGT1A1*28 (TA)7, UGT1A1*36 (TA)5 e UGT1A1*37 (TA)8 della regione promotrice del gene UGT1A1, associati alla tossicità al trattamento con irinotecano, mediante un’unica miscela di oligonucleotidi. La mix UGT1A1 contiene sonde marcate con HEX per il rilevamento di UGT1A1*28 e UGT1A1*37 e sonde marcate con FAM per il rilevamento di UGT1A1*1 e UGT1A1*36.
Controlli
- UGT1A1 WT positive control: Controllo positivo di amplificazione contenente DNA sintetico con genotipo wild-type UGT1A1*1/*1;
- UGT1A1 MT positive control: Controllo positivo di amplificazione contenente DNA sintetico con genotipo mutato UGT1A1*28/*28;
- Controllo negativo.
Campione di partenza
l kit permette l’analisi di DNA genomico estratto da sangue intero.
Caratteristiche
Rilevamento delle principali mutazioni dell’esone 2 (codoni 12,13), dell’esone 3 (codone 61) dei geni KRAS, NRAS, HRAS e dei codoni 600 e 601 del gene BRAF. Ogni mix permette la co-amplificazione di uno o più alleli mutati, più un gene di controllo endogeno.
Controlli
- Controllo positivo, contiene una miscela di sequenze di RNA-DNA sintetico che corrispondono alle principali fusioni rilevate dal kit;
- Controllo negativo.
Campione di partenza
DNA da tessuti freschi, congelati, fissati in formalina (FFPE) e campioni citologici.
Caratteristiche
Determinazione quantitativa di frammenti di DNA di dimensioni maggiori di 200 bp. L’analisi quantitativa del DNA estratto dalle feci in associazione al test iFOBT, rappresenta uno strumento utile per migliorare la diagnosi precoce di CRC permettendo di stimare la probabilità di presenza di CRC stesso.
Controlli
- Controllo positivo, DNA genomico a concentrazione nota;
- Standard, DNA genomico a concentrazione nota da diluire per la creazione della curva standard;
- Spike, DNA di controllo da aggiungere nei campioni per la verifica dell’inibizione;
- Controllo negativo.
Campione di partenza
DNA fecale.
Caratteristiche
l’unico kit completo per uso in vitro diagnostico che permette il rilevamento delle mutazioni T790M e C797S (2 varianti) dell’oncogene EGFR mediante Real-Time PCR. La mutazione T790M rappresenta sia il principale meccanismo di resistenza acquisita nei confronti degli inibitori della tirosin-chinasi di EGFR di prima e seconda generazione, sia il target degli EGFR TKI di terza generazione. Mentre la mutazione C797S è stata riconosciuta da numerosi studi come uno dei fattori responsabili della resistenza verso questi ultimi farmaci.
Controlli
- Controllo positivo, contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Campione di partenza
Il kit permette l’analisi di DNA estratto da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e plasma*.
*Reagenti, consumabili e strumenti accessori per estrazione da plasma sono venduti separatamente (vedere linea Helix, codice H8040).
Caratteristiche
Rilevamento delle principali mutazioni del gene IDH1 (codoni 105 e 132) e del gene IDH2 (codoni 140 e 172). Ogni miscela di oligonucleotidi permette la co-amplificazione di uno o più alleli mutati e di un gene di controllo endogeno. Una specifica miscela di oligonucleotidi di controllo consente la valutazione della qualità e della quantità del DNA presente nei campioni.
Controlli
- Controllo positivo, contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Materiale di partenza
DNA da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE).
Caratteristiche
Rilevamento delle traslocazioni cromosomiche che coinvolgono RET/PTC1: CCDC6-RET; RET/PTC2: PRKAR1A-RET; RET/PTC3: NCOA4-RET e PAX8/PPARG. Ogni miscela di oligonucleotidi permette la co-amplificazione di una o più fusioni e di un gene di controllo endogeno.
Controlli
- Controllo positivo, contiene una miscela di sequenze di RNA-DNA sintetico che corrispondono alle principali fusioni rilevate dal kit;
- Controllo negativo.
Materiale di partenza
RNA da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE) e campioni citologici.
Caratteristiche
Rilevamento di 8 marcatori “quasi-monomorfici” mononucleotidici: BAT-25, BAT-26, NR-21, NR-22, NR-24, NR-27, CAT-25 e MONO-27 mediante PCR Real Time e successiva analisi dei target sulla base della curva di denaturazione. Il test permette di rilevare in modo accurato e con ridotto “tempo operatore” l’instabilità microsatellitare in campioni tumorali.
Controlli
- Controllo positivo, DNA genomico corrispondente ad un profilo stabile;
- Controllo negativo.
Materiale di partenza
DNA da tessuti freschi, congelati, fissati in formalina e inclusi in paraffina (FFPE). Non è necessaria la comparazione con tessuto normale o sangue per l’analisi dei risultati.
Caratteristiche
Identificazione di 14 genotipi High Risk (16, 18, 31, 33, 35, 39, 45, 51, 52, 56, 58, 59, 66 e 68) dello Human Papilloma Virus (HPV) tramite l’amplificazione degli oncogeni E6 ed E7. Ogni mix permette la co-amplificazione con sonde genotipo-specifiche di target HPV e di un gene di controllo endogeno.
Materiale di partenza
DNA da tamponi cervicali e da tessuti fissati in formalina e inclusi in paraffina (FFPE).
Caratteristiche
Rilevamento delle varianti di fusione dei geni NTRK1, NTRK2 ed NTRK3 mediante One Step Real Time. Il test permette di rilevare la presenza dei riarrangiamenti genici identificati dal kit mediante l’utilizzo di primer e probe che permettono di amplificare nel canale FAM in modo selettivo le fusioni analizzate dal kit.
Controlli
- Saggio di controllo: è presente un saggio specifico per la valutazione del contenuto di DNA nel campione. Inoltre, in ogni mix viene rilevato un gene di controllo endogeno che permette di verificare la corretta esecuzione della procedura di amplificazione e l’eventuale presenza di inibitori, che possono causare dei risultati falsamente negativi;
- Controllo positivo: contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Materiale di partenza
RNA isolato da tessuti freschi, congelati, fissati in formalina, inclusi in paraffina (FFPE) e campioni citologici.
Caratteristiche
Rilevamento delle principali mutazioni del gene PIK3CA (Codoni 345, 420, 542, 545, 546, 1047, 1049).
Controlli
- Saggio di controllo: è presente un saggio specifico per la valutazione del contenuto di DNA nel campione. Inoltre in ogni mix viene rilevato un gene di controllo endogeno che permette di verificare la corretta esecuzione della procedura di amplificazione e l’eventuale presenza di inibitori, che possono causare dei risultati falsamente negativi;
- Controllo positivo: contiene una miscela di sequenze di DNA sintetico che corrispondono ad ogni mutazione rilevata dal kit e di DNA genomico wild-type;
- Controllo negativo.
Materiale di partenza
DNA genomico isolato da tessuti freschi, congelati, fissati in formalina, inclusi in paraffina (FFPE) o da plasma*.
*Reagenti, consumabili e strumenti accessori per estrazione da plasma sono venduti separatamente (vedere linea Helix, codice H8040).
Caratteristiche
Rilevamento qualitativo mediante RT-PCR dello stato di metilazione di 12 isole CpG collocate nel promotore del gene MGMT. Il kit comprende i reagenti per il trattamento al bisolfito di sodio del DNA estratto prima dell’analisi di metilazione, il quale converte le citosine non metilate in uracile.
Controlli
- Controllo positivo metilato.
- Controllo positivo non metilato.
- Controllo negativo.
Materiale di partenza
DNA da tessuti fissati in formalina e inclusi in paraffina (FFPE).
Caratteristiche
Rilevamento delle più frequenti mutazioni del gene FGFR3 negli esoni 7, 9, 14 e delle fusioni dei geni FGFR2, FGFR3, mediante 8 miscele di oligonucleotidi. Ogni miscela di oligonucleotidi permette la co-amplificazione del target e di un gene di controllo endogeno.
Una specifica miscela di oligonucleotidi di controllo consente la valutazione della qualità e della quantità di ognicampione.
Controlli
- Controllo positivo, contenente una miscela di sequenze di DNA sintetico wild – type che corrisponde ad ogni variante rilevata dal kit, in un background di DNA ed RNA wild type.
- Controllo negativo.
Sensibilità
Il kit ha dimostrato una specificità del 100% e una sensibilità superiore al 97%.
Campione di partenza
Il kit permette l’analisi di DNA ed RNA estratto da tessuti freschi, congelati, fissati in formalina inclusi in paraffina (FFPE).

Seleziona una o più brochure e inserisci i tuoi dati per procedere al download
Categoria
Realtime PCR




